Ich habe mir letztens, aus gegebenen Anlass, die wunderbare Weihnachtsvorlesung von Dr. Edmund Weitz angesehen und bin gegen Ende im dritten Teil stecken geblieben.
Die Poincaré-Vermutung (Teil 3 von 3, Weihnachtsvorlesung 2018)
Es wurde die Frage gestellt, Wie sieht das Leben auf einem Torus aus?
Ich mach es kurz: Wenn das Universum ein Torus wäre (in höherer Dimension versteht sich), dann wäre es endlich, ohne Rand. Man könnte in eine Richtung schauen und würde irgendwann sich selbst von Hinten sehen. Und wenn man noch weiter schaut, sieht man sich selbst noch einmal, und noch einmal u.s.w. Natürlich ist die Sicht immer etwas verzerrt.
Damit ergibt auch folgender Satz Sinn: Das Universum ist kleiner als das was wir sehen können. Bzw.: Das beobachtbare Universum ist größer als das eigentliche.
Wie auch immer, zur Veranschaulichung wurde in 2D einige Punkte erstellt, nach rechts/links/oben/unten kopiert und von jedem Punkt zu jedem anderen den Abstand ermittelt und davon ein Histogramm erzeugt. Sieht etwa bei Minute 31:30.
Der Witz ist nun folgender: Gibt es keine sich wiederholende Muster, ist eine Poison Verteilung sichtbar. Gibt es solche Muster, hat die Verteilung Peaks.
Na, das lässt sich doch wunderbar auf ein Digitales Geländemodell (DGM) anwenden!
In der Regel sind die Punkte aus unstrukturierte Laserscans ziemlich dicht verteilt. Es gibt Bereiche an denen sich mehr Messpunkte befinden. Das Flugzeug fliegt die Landschaft ja hin und her ab, und da gibt es Überlappungen. Auch bei der ersten Nachbearbeitung der Daten passieren magische Dinge. Ich habe also keine Ahnung wie das Histogramm aussieht und bin sehr gespannt.
Als Datensatz fand ich von Open Geodata NRW viele DGMs. Die kleinste Datei dgm1l_05974056_Wickede_Ruhr_EPSG25832_XYZ.zip beinhaltete aber immer noch viele GB an Daten. Also nutze ich nur die Kachel 32420_5705 mit 1812468 (1.8M) Punkten.
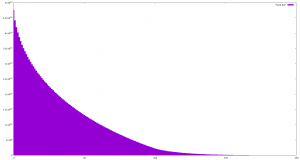
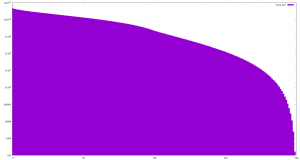
So sieht die Höhenverteilung aus:

Und hier die Punktverteilung. Nur an einigen weißen Stellen sind nicht so viele Punkte vorhanden.

Als nächstes schrieb ich ein Programm welches die Punkte einliest, in einer quadratischen Schleife alle Punkt Abstände berechnet und on-thy-fly ein Histogramm mit den ersten drei Momenten erzeugt. Dabei werden die Abstände zweier Punkte nur einmal berechnet. Also von Punkt A zu B. Aber nicht von B zu A. Der ist ja der selbe. Damit ergeben sich 1642 Milliarden Abstandsberechnungen. Um die alle zu speichern, bräuchte man 12233 GB an Speicherplatz. Den habe ich nicht. Daher habe ich ein Histogramm entwickelt, welches kontinuierlich mit Daten erstellt werden kann.
Auch die Laufzeit ist bei diesen Algorithmus ein Problem. Also berechne ich nur den quadratischen Abstand. Das spart eine Wurzel Berechnung. Und in der Summe ist das schon was. Zusätzlich gibt es noch ein Point2D mit SIMD/SSE Fähigkeiten. Dies spart noch ein paar Operationen pro Abstands Berechnung.
Auf einem 4GHz CPU dauerte die Berechnung drei Stunden. Für einen doppelt so großen Datensatz sind es schon 9 Stunden. Und für nicht unübliche 100M Punkte würde es ca. 12 Monate dauern. Immerhin noch realistisch.
Aber wie sieht nun das Histogramm aus? Nicht vergessen: Das sind quadratische Abstände. In Klammern die Wurzel aus den Werten.
Min: 0 Avg: 3.31e+05 (575.3) Max: 2e+06 (1414.2) Var: 7.71e+10 (277668.9) Std 2.78e+05 (527.3)
Der minimaler Abstand von 0 deutet auf doppelte Punkte hin. Das ist nicht ungewöhnlich.
Der maximale Abstand ist gerade die Länge der Diagonalen der Boundingbox von jeweils 1km Seitenlänge. Auch richtig.
Der mittlere Abstand ist auch okay. Stelle ich mich gedanklich in die Mitte der BB, habe ich nach allen Seiten mehr als 500m Platz.
Keine Peaks zu erkennen. Alles gut. Oder das Histogramm ist nicht fein genug ;)
Update:
Auch bei einem Histogramm mit 5000 Bins zeigen sich keine Peaks.